Servizi
|
Identificazione di proteine (in-gel)
Il workflow include digestione in-gel, purificazione dei peptidi mediante StageTips (Rappsilber et al, Nat. Prot. 2007), analisi LC-MS/MS (gradiente di 45 min) e ricerca in banca dati (Sequest search engine, Proteome Discoverer 2.1). Risultati attesi: identificazione di qualsiasi proteina rilevabile mediante colorazione d'argento o Coomassie. Output: Lista di proteine identificate (min 2 peptidi al 99% di confidenza), file mgf su richiesta. |
|
Identificazione di proteine (in-soluzione)
Se le condizioni sperimentali lo permettono, la digestione in soluzione è l'approccio più immediato per identificare una proteina purificata. Il workflow comprende digestione in soluzione, purificazione di peptidi mediante StageTips (Rappsilber et al, Nat. Prot. 2007), analisi LC-MS/MS (gradiente di 45 min) e ricerca in banca dati (Sequest search engine, Proteome Discoverer 2.1). Risultati attesi: identificazione di proteine presenti in quantità superiori ad 1 ng. Output: Lista di proteine identificate (min 2 peptidi al 99% di confidenza), file mgf su richiesta. |
|
Bottom-up proteomics (qualitativa)
Sebbene in genere lo scopo di un'analisi proteomica sia la determinazione quantitativa delle proteine costituenti la miscela, in alcuni casi una semplice lista delle proteine rilevabili in un campione può essere un'informazione importante. Il workflow comprende precipitazione delle proteine, digestione in soluzione, purificazione dei peptidi mediante StageTips (Rappsilber et al, Nat. Prot. 2007), analisi LC-MS/MS (gradiente di 90 min) e ricerca in banca dati (Sequest search engine, Proteome Discoverer 2.1). Risultati attesi: Identificazione di 1500-2500 proteine (da lista cellulare) da un minimo di 1 microgrammo di proteine totali. Nel caso si includa un frazionamento di peptidi (n=6), il numero di identificazioni può arrivare a 5000. Output: Lista delle proteine identificate (min 2 peptidi al 99% di confidenza) file mgf su richiesta. |
|
Analisi proteomica quantitativa
L'analisi proteomica quantitativa può basarsi su metodi di marcatura isotopica (TMT, dimethyl labeling, SILAC) o su approcci label-free. Il workflow da noi suggerito consiste nei seguenti passaggi: (i) digestione delle proteine; (ii) analisi nLC-MS/MS (gradiente maggiore di 120 min); (iii) ricerca in banca dati e quantificazione label-free (Maxquant software). Su richiesta, è effettuabile analisi quantitativa mediante marcatura isotopica (TMT, dimethyl labeling, SILAC). Risultati attesi: quantificazione di almeno 3000 proteine basata su almeno un peptide unico (protein FDR inferiore a 1%). Output: Lista di proteine identificate e quantificate. Dati sui peptidi. |
|
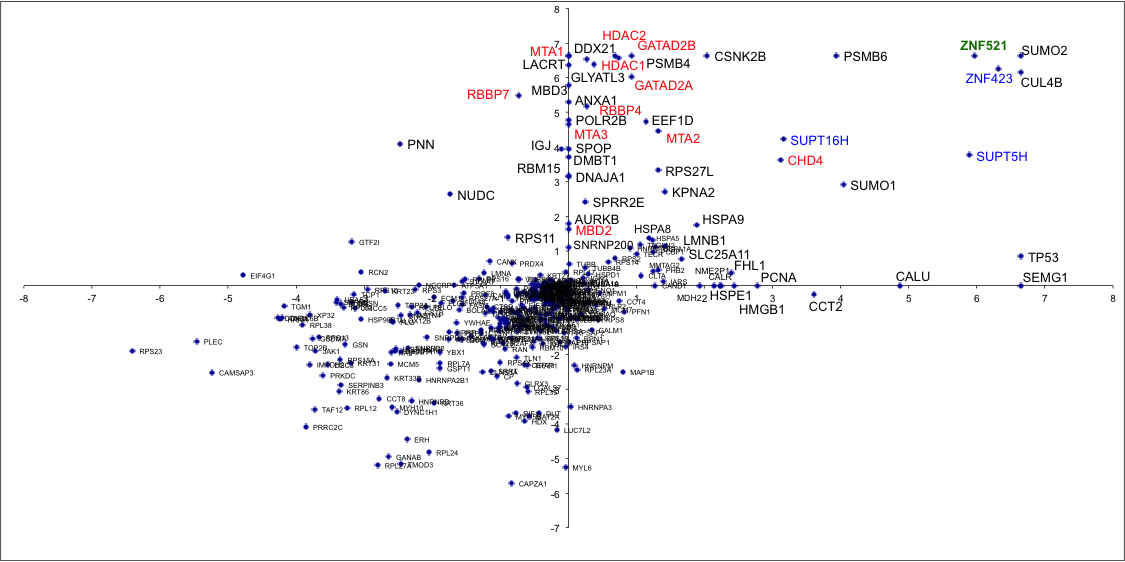
Studio di interazioni proteina-proteina
Affinity enrichment- Mass spectrometry (AE-MS) è diventato un approccio essenziale per lo studio di complessi proteici. Il nostro laboratorio può effettuare analisi quantitativa comparativa tra un immunoprecipitato di proteine di interesse e campioni-controllo scelti opportunamente. Il workflow si basa su digestione on-beads, ed analisi nLC-MS/MS in modalità label-free. Risultati attesi: identificazione di 1000 proteine (la maggioranza delle quali, dovute ad interazioni a-specifiche con l'anticorpo); rilevamento di (generalmente) 10-50 interattori. Output: Identificazione di proteine e tabelle quantitative, contenenti informazioni sulle proteine significativamente arricchite. |